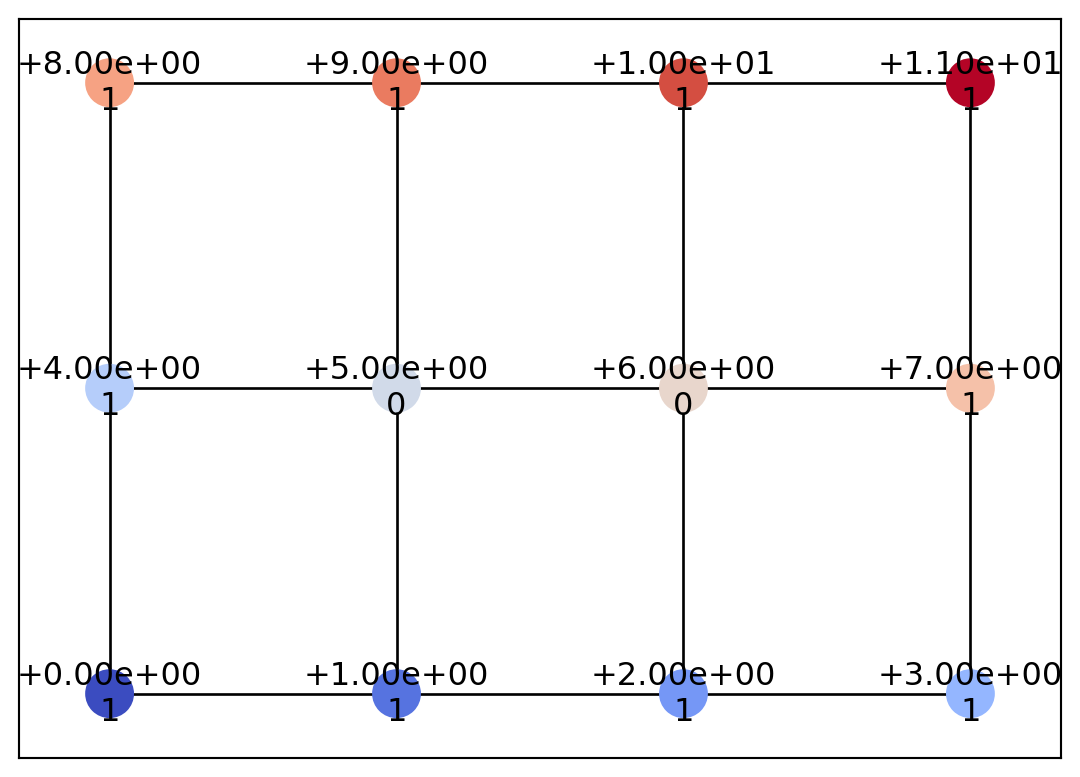
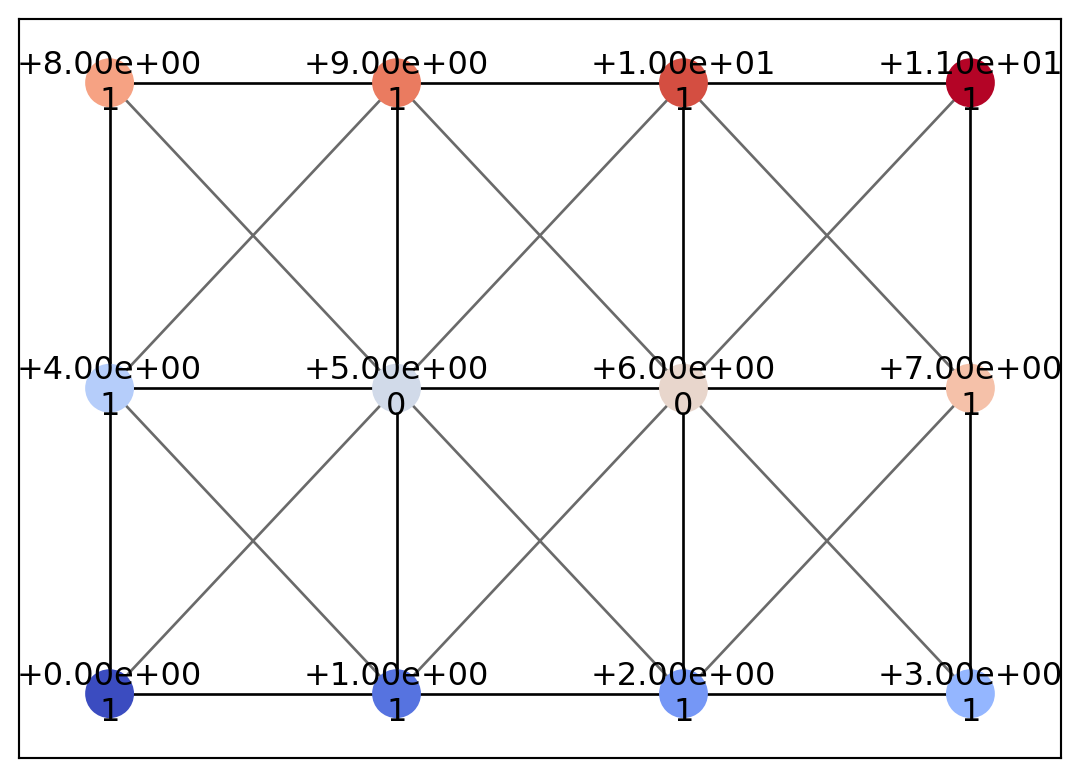
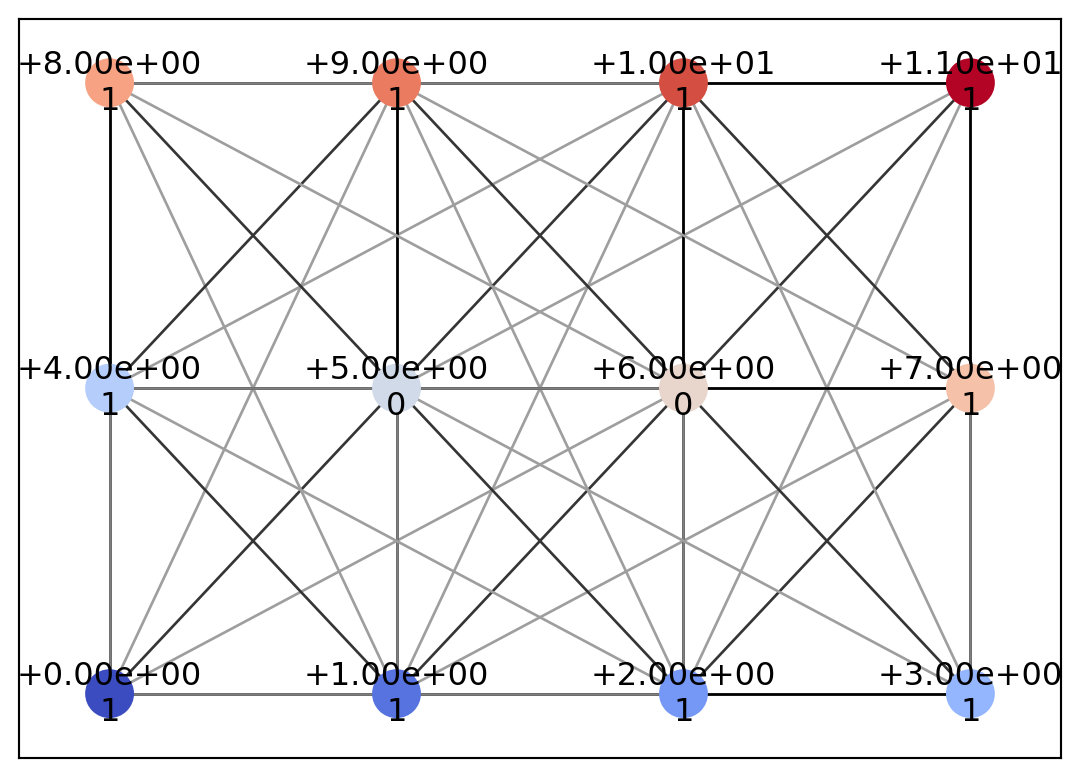
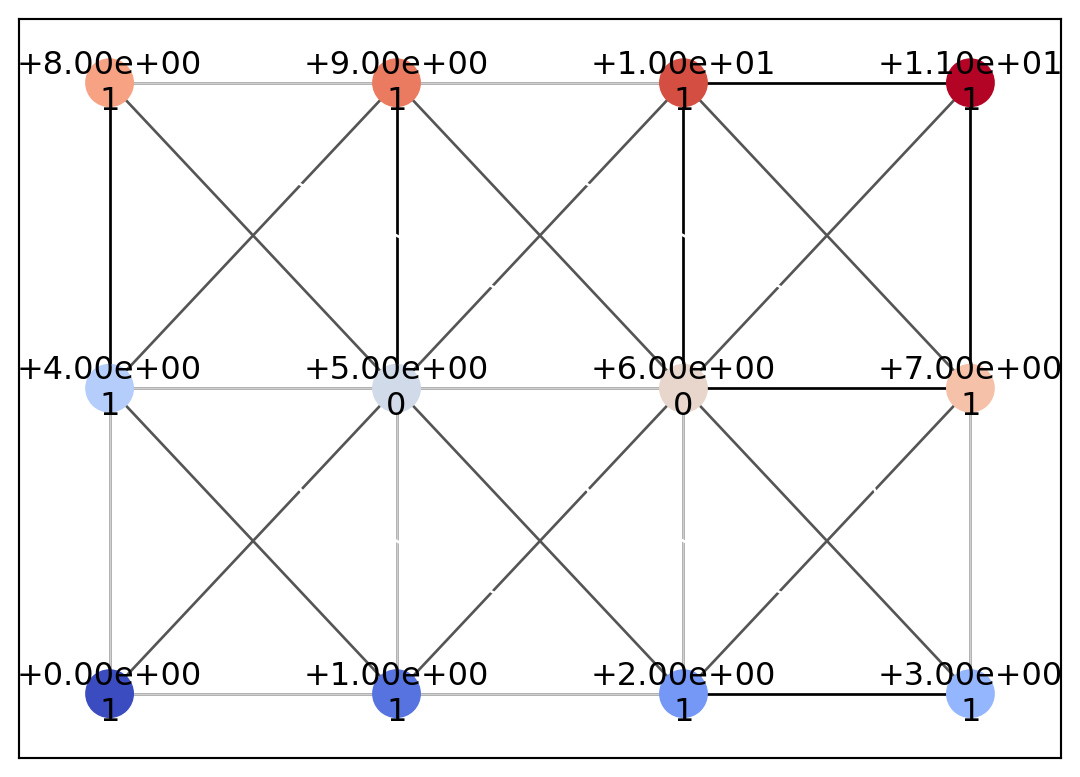
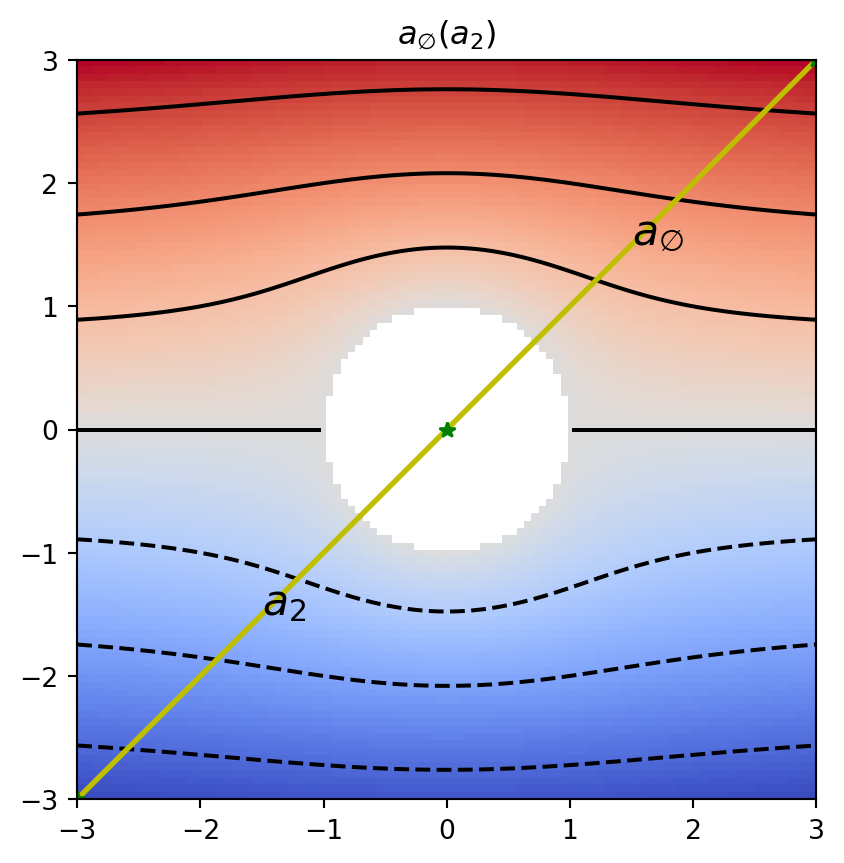
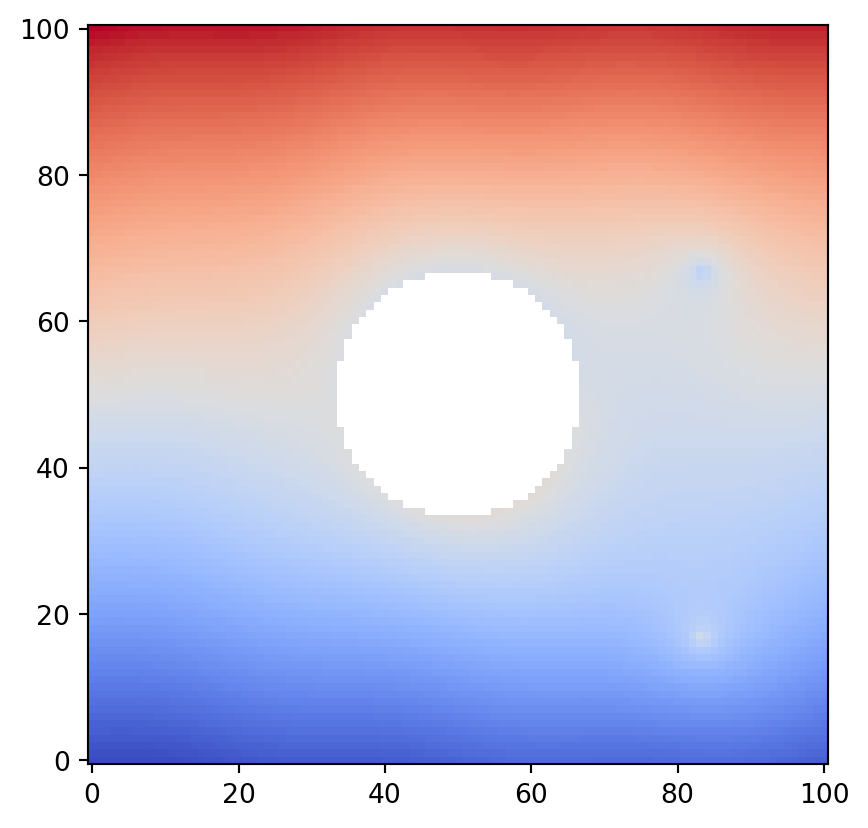
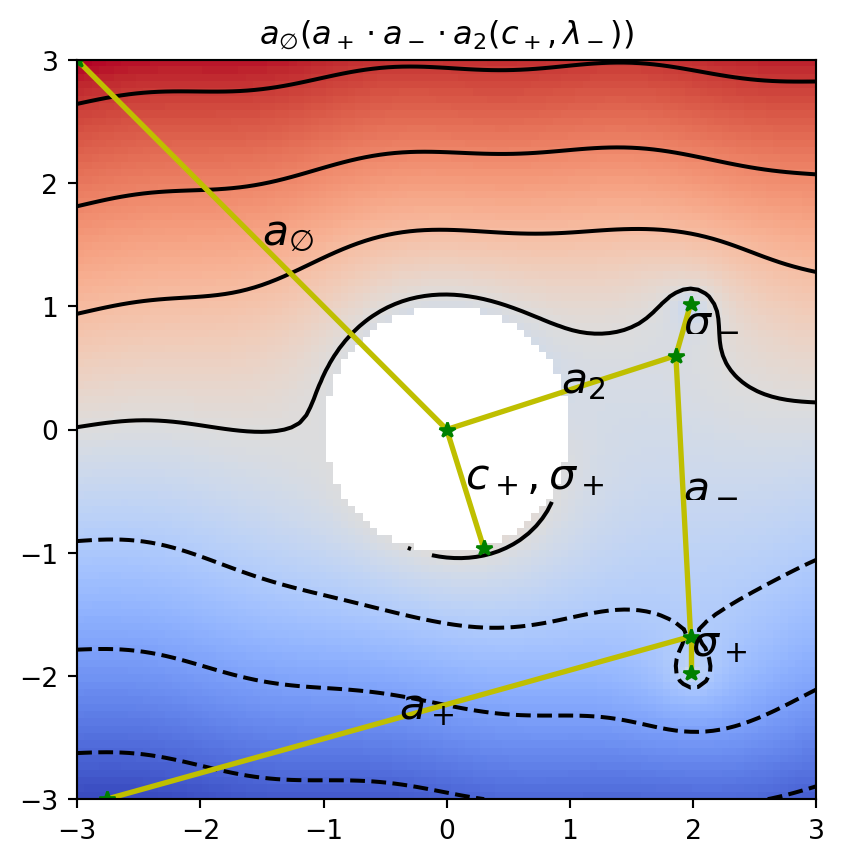
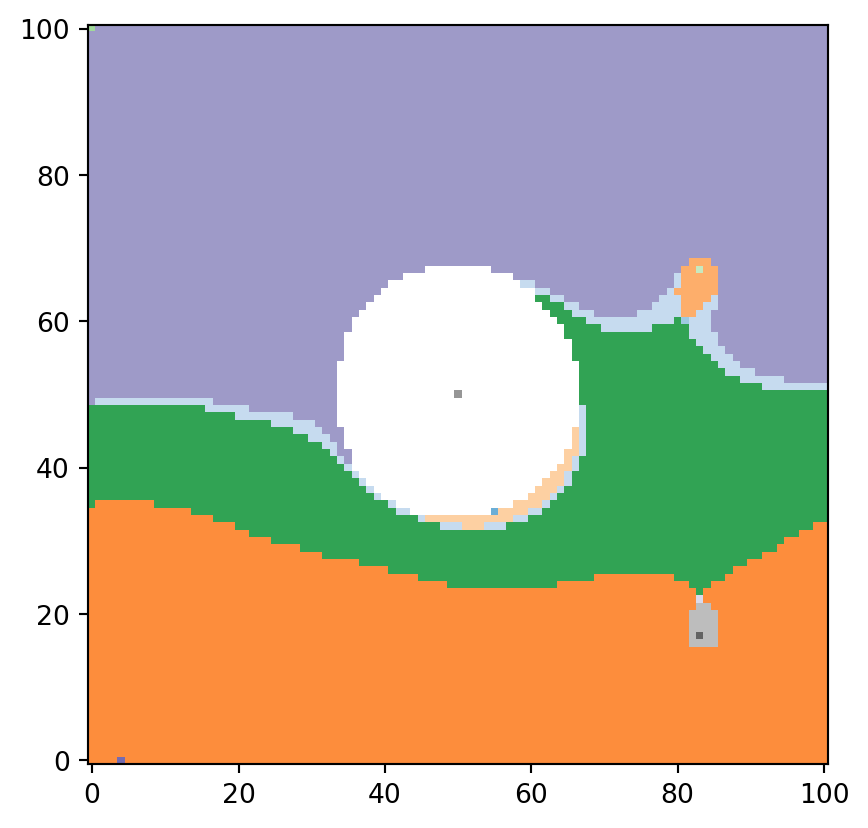
{Point(id=np.int64(0), level=np.int64(0), x=np.int64(0), y=np.int64(0), geotype=np.int64(1), order=np.float64(-2.498091544796509)): [<psiclone.data.graph.Edge at 0x7fb641715250>,
<psiclone.data.graph.Edge at 0x7fb641717410>,
<psiclone.data.graph.Edge at 0x7fb641715670>,
<psiclone.data.graph.Edge at 0x7fb6417148f0>,
<psiclone.data.graph.Edge at 0x7fb641744a40>,
<psiclone.data.graph.Edge at 0x7fb641746360>,
<psiclone.data.graph.Edge at 0x7fb6417459a0>],
Point(id=np.int64(1), level=np.int64(1), x=np.int64(1), y=np.int64(0), geotype=np.int64(1), order=np.float64(-2.1587989303424644)): [<psiclone.data.graph.Edge at 0x7fb641715250>,
<psiclone.data.graph.Edge at 0x7fb641716000>,
<psiclone.data.graph.Edge at 0x7fb641745130>,
<psiclone.data.graph.Edge at 0x7fb641744bf0>,
<psiclone.data.graph.Edge at 0x7fb641744da0>,
<psiclone.data.graph.Edge at 0x7fb641744680>,
<psiclone.data.graph.Edge at 0x7fb641745b80>,
<psiclone.data.graph.Edge at 0x7fb641744230>,
<psiclone.data.graph.Edge at 0x7fb6417447d0>,
<psiclone.data.graph.Edge at 0x7fb641744e30>],
Point(id=np.int64(2), level=np.int64(2), x=np.int64(2), y=np.int64(0), geotype=np.int64(1), order=np.float64(-1.5707963267948966)): [<psiclone.data.graph.Edge at 0x7fb641717410>,
<psiclone.data.graph.Edge at 0x7fb641716000>,
<psiclone.data.graph.Edge at 0x7fb6417468a0>,
<psiclone.data.graph.Edge at 0x7fb641745a30>,
<psiclone.data.graph.Edge at 0x7fb6417456a0>,
<psiclone.data.graph.Edge at 0x7fb641745af0>,
<psiclone.data.graph.Edge at 0x7fb641746660>,
<psiclone.data.graph.Edge at 0x7fb641746600>,
<psiclone.data.graph.Edge at 0x7fb641745880>,
<psiclone.data.graph.Edge at 0x7fb64175f8f0>],
Point(id=np.int64(3), level=np.int64(3), x=np.int64(3), y=np.int64(0), geotype=np.int64(1), order=np.float64(-0.982793723247329)): [<psiclone.data.graph.Edge at 0x7fb641745130>,
<psiclone.data.graph.Edge at 0x7fb6417468a0>,
<psiclone.data.graph.Edge at 0x7fb641745dc0>,
<psiclone.data.graph.Edge at 0x7fb64175fc50>,
<psiclone.data.graph.Edge at 0x7fb64175ec00>,
<psiclone.data.graph.Edge at 0x7fb64175e9c0>,
<psiclone.data.graph.Edge at 0x7fb64175f230>],
Point(id=np.int64(4), level=np.int64(4), x=np.int64(0), y=np.int64(1), geotype=np.int64(1), order=np.float64(-2.896613990462929)): [<psiclone.data.graph.Edge at 0x7fb641715670>,
<psiclone.data.graph.Edge at 0x7fb641744bf0>,
<psiclone.data.graph.Edge at 0x7fb641745a30>,
<psiclone.data.graph.Edge at 0x7fb64175e150>,
<psiclone.data.graph.Edge at 0x7fb64175dfd0>,
<psiclone.data.graph.Edge at 0x7fb64175d310>,
<psiclone.data.graph.Edge at 0x7fb64175d130>,
<psiclone.data.graph.Edge at 0x7fb64175d430>],
Point(id=np.int64(5), level=np.int64(5), x=np.int64(1), y=np.int64(1), geotype=np.int64(0), order=np.float64(nan)): [<psiclone.data.graph.Edge at 0x7fb6417148f0>,
<psiclone.data.graph.Edge at 0x7fb641744da0>,
<psiclone.data.graph.Edge at 0x7fb6417456a0>,
<psiclone.data.graph.Edge at 0x7fb641745dc0>,
<psiclone.data.graph.Edge at 0x7fb64175e150>,
<psiclone.data.graph.Edge at 0x7fb64175e360>,
<psiclone.data.graph.Edge at 0x7fb64175f8c0>,
<psiclone.data.graph.Edge at 0x7fb64175dd30>,
<psiclone.data.graph.Edge at 0x7fb64175c260>,
<psiclone.data.graph.Edge at 0x7fb64175d250>,
<psiclone.data.graph.Edge at 0x7fb64175f050>],
Point(id=np.int64(6), level=np.int64(6), x=np.int64(2), y=np.int64(1), geotype=np.int64(0), order=np.float64(nan)): [<psiclone.data.graph.Edge at 0x7fb641744a40>,
<psiclone.data.graph.Edge at 0x7fb641744680>,
<psiclone.data.graph.Edge at 0x7fb641745af0>,
<psiclone.data.graph.Edge at 0x7fb64175fc50>,
<psiclone.data.graph.Edge at 0x7fb64175dfd0>,
<psiclone.data.graph.Edge at 0x7fb64175e360>,
<psiclone.data.graph.Edge at 0x7fb64175e390>,
<psiclone.data.graph.Edge at 0x7fb64175ce90>,
<psiclone.data.graph.Edge at 0x7fb64175c890>,
<psiclone.data.graph.Edge at 0x7fb641857500>,
<psiclone.data.graph.Edge at 0x7fb641854920>],
Point(id=np.int64(7), level=np.int64(7), x=np.int64(3), y=np.int64(1), geotype=np.int64(1), order=np.float64(-0.4636476090008061)): [<psiclone.data.graph.Edge at 0x7fb641745b80>,
<psiclone.data.graph.Edge at 0x7fb641746660>,
<psiclone.data.graph.Edge at 0x7fb64175ec00>,
<psiclone.data.graph.Edge at 0x7fb64175f8c0>,
<psiclone.data.graph.Edge at 0x7fb64175e390>,
<psiclone.data.graph.Edge at 0x7fb64175e1b0>,
<psiclone.data.graph.Edge at 0x7fb6418544a0>,
<psiclone.data.graph.Edge at 0x7fb641856b40>],
Point(id=np.int64(8), level=np.int64(8), x=np.int64(0), y=np.int64(2), geotype=np.int64(1), order=np.float64(2.896613990462929)): [<psiclone.data.graph.Edge at 0x7fb641746360>,
<psiclone.data.graph.Edge at 0x7fb641744230>,
<psiclone.data.graph.Edge at 0x7fb64175d310>,
<psiclone.data.graph.Edge at 0x7fb64175dd30>,
<psiclone.data.graph.Edge at 0x7fb64175ce90>,
<psiclone.data.graph.Edge at 0x7fb6418569c0>,
<psiclone.data.graph.Edge at 0x7fb6418546e0>],
Point(id=np.int64(9), level=np.int64(9), x=np.int64(1), y=np.int64(2), geotype=np.int64(1), order=np.float64(2.677945044588987)): [<psiclone.data.graph.Edge at 0x7fb6417459a0>,
<psiclone.data.graph.Edge at 0x7fb6417447d0>,
<psiclone.data.graph.Edge at 0x7fb641746600>,
<psiclone.data.graph.Edge at 0x7fb64175d130>,
<psiclone.data.graph.Edge at 0x7fb64175c260>,
<psiclone.data.graph.Edge at 0x7fb64175c890>,
<psiclone.data.graph.Edge at 0x7fb64175e1b0>,
<psiclone.data.graph.Edge at 0x7fb6418569c0>,
<psiclone.data.graph.Edge at 0x7fb641854770>,
<psiclone.data.graph.Edge at 0x7fb64187e420>],
Point(id=np.int64(10), level=np.int64(10), x=np.int64(2), y=np.int64(2), geotype=np.int64(1), order=np.float64(1.5707963267948966)): [<psiclone.data.graph.Edge at 0x7fb641744e30>,
<psiclone.data.graph.Edge at 0x7fb641745880>,
<psiclone.data.graph.Edge at 0x7fb64175e9c0>,
<psiclone.data.graph.Edge at 0x7fb64175d430>,
<psiclone.data.graph.Edge at 0x7fb64175d250>,
<psiclone.data.graph.Edge at 0x7fb641857500>,
<psiclone.data.graph.Edge at 0x7fb6418544a0>,
<psiclone.data.graph.Edge at 0x7fb6418546e0>,
<psiclone.data.graph.Edge at 0x7fb641854770>,
<psiclone.data.graph.Edge at 0x7fb641856480>],
Point(id=np.int64(11), level=np.int64(11), x=np.int64(3), y=np.int64(2), geotype=np.int64(1), order=np.float64(0.4636476090008061)): [<psiclone.data.graph.Edge at 0x7fb64175f8f0>,
<psiclone.data.graph.Edge at 0x7fb64175f230>,
<psiclone.data.graph.Edge at 0x7fb64175f050>,
<psiclone.data.graph.Edge at 0x7fb641854920>,
<psiclone.data.graph.Edge at 0x7fb641856b40>,
<psiclone.data.graph.Edge at 0x7fb64187e420>,
<psiclone.data.graph.Edge at 0x7fb641856480>]}